Pesquisadores do LNCC desenvolvem plataforma para ajudar na busca de tratamentos da Covid-19
Portal web faz uma triagem virtual para a busca de medicamentos contra o coronavírus e é disponibilizado por pesquisadores do LNCC à comunidade científica.
A plataforma computacional DockThor-VS (https://www.dockthor.lncc.br), utilizada amplamente para a pesquisa de novos medicamentos de forma virtual, foi desenvolvida em 2013 pelo Grupo de Modelagem Molecular em Sistemas Biológicos (GMMSB) do Laboratório Nacional de Computação Científica e está acoplada ao supercomputador Santos Dumont. Diante da pandemia da Covid-19, ela foi incrementada para disponibilizar aos usuários estruturas tridimensionais de proteínas-alvo importantes para o desenvolvimento de novas moléculas e reposicionamento de fármacos para o tratamento da doença.
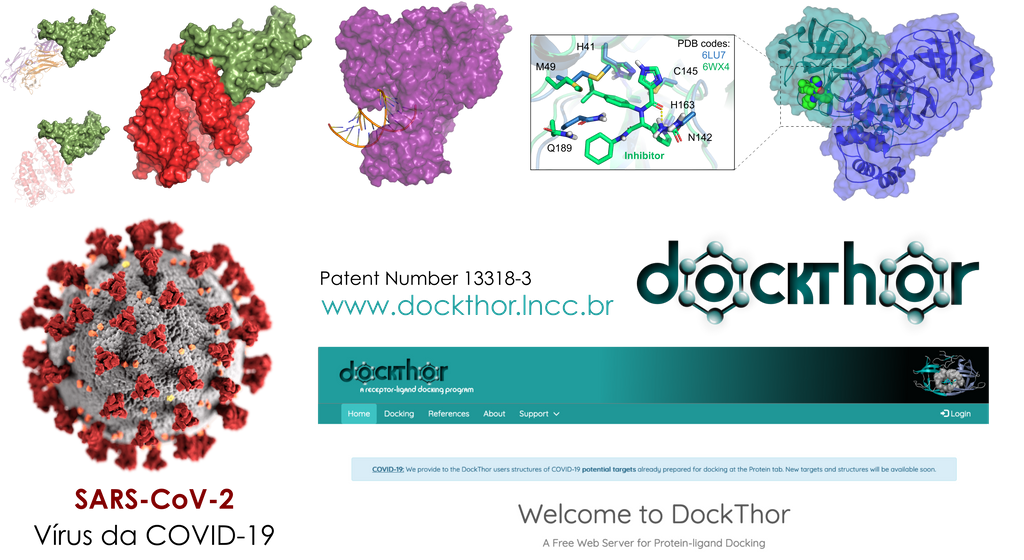
Dentre os aspectos mais relevantes associados ao desenvolvimento desta plataforma, estão a disponibilização de diferentes “estados conformacionais” de cada proteína alvo, simulando sua flexibilidade em meio biológico; modelagem tridimensional de mutações relevantes destas proteínas que podem afetar a ação de possíveis fármacos e gerar resistência a medicamentos, identificadas a partir da análise de milhares de genomas de SARS-CoV-2 sequenciados no mundo todo; e disponibilização de banco de estruturas tridimensionais dos fármacos já aprovados pelo Food and Drug Administration (FDA) para estudos de reposicionamento de fármacos.
Essa estratégia tem sido amplamente utilizada em todo o mundo e tem como objetivo identificar fármacos utilizados atualmente para o tratamento de outras doenças que poderiam atuar também no tratamento da Covid-19.
O desenvolvimento do DockThor-VS direcionado para alvos terapêuticos do vírus SARS-CoV-2 foi coordenado pelo prof. Laurent Dardenne (LNCC) em colaboração com a Profa. Marisa Nicolás (LNCC), a qual coordenou os estudos genômicos dos alvos terapêuticos. O desenvolvimento da plataforma, incluindo as técnicas de inteligência computacional para triagem virtual, foi inteiramente realizado por pesquisadores do LNCC, incluindo a pesquisadora de pós-doutorado PCI/MCTI Isabella Guedes, alunos e ex-alunos da Pós-Graduação em Modelagem Computacional do LNCC.
“A combinação e disponibilização de informações estruturais sobre os alvos terapêuticos, técnicas computacionais avançadas para o planejamento de fármacos e recursos computacionais de alto desempenho podem direcionar e acelerar as pesquisas de diversos grupos brasileiros e internacionais na procura por um tratamento para Covid-19”, destaca Laurent Dardenne. “Disponibilizar para a comunidade científica as variantes genômicas das proteínas do vírus que são alvos terapêuticos é muito importante para desenvolver medicamentos, vacinas e testes diagnósticos mais eficazes, contornando problemas como resistência a fármacos antivirais”, ressalta Marisa Nicolás.

Pesquisadores do LNCC responsáveis pelo desenvolvimento da plataforma. Foto Divulgação
Um artigo científico será submetido em breve para revista indexada internacional contendo os detalhes de implementação da plataforma DockThor-VS e os resultados de estudos computacionais de reposicionamento de fármacos para todas as proteínas alvo do SARS-CoV-2 disponibilizadas. Atualmente, o portal possui 35 estruturas tridimensionais relacionadas a seis diferentes proteínas-alvo do SARS-CoV-2.
A plataforma DockThor-VS está disponível de forma gratuita para pesquisadores de todo o mundo. Os usuários não cadastrados podem realizar experimentos com até 200 moléculas por vez, enquanto que estudos mais complexos envolvendo até 5 mil moléculas estão liberados para usuários com projetos submetidos e aprovados. A plataforma está totalmente operacional, tendo sido submetidos nos primeiros quinze dias de agosto cerca de 1200 experimentos de triagem virtual.

Foto Divulgação LNCC
O trabalho foi realizado com financiamento das agências de fomento à pesquisa FAPERJ, CAPES e CNPq.
Veja também:
















